COMPARACIÓN DE TRES MÉTODOS DE EXTRACCIÓN DE ADN DE MUESTRAS DE PIEL Y PELO DE JAGUAR (Panthera onca)
COMPARISON OF THREE METHODS FOR DNA EXTRATIONS FROM SKIN AND HAIR SAMPLES OF JAGUAR (Panthera onca)
Janeth Ortega Torres
Universidad Nacional Abierta y a Distancia; Fundación IASSOS
myriam.ortega@unad.edu.co
Engelberth Chávez Fontecha>
Fundación IASSOS
José Camilo Torres Romero
Universidad Nacional Abierta y a Distancia
jose.torres@unad.edu.co
Jessica Braga
Universidad Nacional Abierta y a Distancia
jessica.almeida@unad.edu.co
Resumen
El jaguar americano es una de las especies de felinos que habita en todo el continente americano. En Colombia, su distribución geográfica lo sitúa desde la Sierra Nevada de Santa Marta hasta el Amazonas. Diferentes factores antrópicos amenazan la persistencia de la especie, la cual se encuentra dentro de la lista roja CITIES, como especie amenazada. Análisis moleculares de especies, utilizando diversos marcadores de ADN, permiten discernir subespecies, filogeografía, diversidad genética y patrones de parentesco. El objetivo de este trabajo fue obtener DNA de buena calidad de muestras antiguas. Se analizaron fragmentos de ADN de tejido antiguo de jaguar (14 años) obtenido de fragmentos de piel sin tratamiento de preservación y se probaron tres protocolos de extracción de ADN y amplificó un segmento del gen mitocondrial ATP6-DF3. Los protocolos utilizados fueron: extracción por resina quelante-Quelex, Fenol–cloroformo y Kit de extracción comercial. Los resultados indican que en todos las muestras de fragmentos de piel de jaguar, se obtuvo ADN, a pesar de su antigüedad. Los métodos de extracción que presentaron mejores resultados fueron el que utilizó el kit comercial y fenol cloroformo, además el método de quelex mostró ser util, rápido y barato para obtener ADN de suficiente calidad, en muetras difícles, por su antigüedad y poco estado de preservación.
Palabras claves: conservación; ADN; jaguar.
Abstract
American jaguar is one of the largest felids that inhabits the American continent. In Colombia, their geographical distributions ranging from Sierra Nevada of Santa Marta to Amazonas. Different anthropic factors threaten the persistence of the species, which is Near Theratened on the CITIES red list. Analysis at the molecular level of the species, using different DNA markers, is increasing and necessary to study subspecies, phylogeography, genetic diversity and kinship patterns. The aims of this work was to obtain good quality DNA from old tissue (14 years), which was obtained from skin fragments without preservation treatment. Here we evaluated the potencial of three diferents protocols of DNA extraction and amplification of a segment of the mitochondrial ATP6-DF3. Extration protocols used were: extraction by chelating resin-Quelex, phenol-chloroform and commercial extraction kit. Results indicate that in all samples of jaguar skin fragments, DNA was obtained, despite its age. The extraction methods applied to differents skin frgaments, results in DNA of sufficient amont and purity when a kit and phenol –cloroform were used. In addition, quelex procedure is proved to be simple, rapid and unexpensive to obtain DNA of sufficient quality, from difficult samples due to its antiquity and poor state of preservation.
Key words: conservation; DNA; jaguar.
Introducción
Los estudios de ADN son una herramienta importante para el análisis de la diversidad biológica y genética de las especies, además permiten definir patrones biogeográficos, filogenéticos y relaciones de parentesco entre los individuos. Muestras de pelos, plumas, heces, orina, cáscaras de huevo, escamas, tejido blando y duro de animales muertos, especímenes de museo y hasta fósiles, son potenciales fuentes de ADN (Beja-Pereira et al., 2009).
El jaguar americano (Phantera onca) se encuentra clasificado en el apéndice I de CITIES, en la lista roja como en peligro de extinción, debido a que en los pasados 21 años, declinó su población en cerca del 25%. En Colombia, la especie ocupa todo el territorio nacional, sin embargo, debido a la deforestación de su hábitat y caza indiscriminada el tamaño poblacional es cada vez menor. A pesar de ello, la especie reporta altos valores de diversidad genética (heterocigosidad 0.738) (Ruiz et al., 2006), lo que es promisorio para la conservación de la misma, la cual sigue necesitando esfuerzos e investigación para proponer mejores rutas de conservación.
Una de las principales herramientas moleculares utilizadas para amplificar regiones especificadas de ADN, a partir de muestras de diferente origen, grado de conservación y antigüedad es la PCR (reacción en cadena de la polimerasa), sin embargo, esta técnica puede ser problemática cuando se parte de ADN obtenido de muestras llamadas difíciles, ya que pueden contener diferentes tipos de inhibidores de la reacción, ADN degradado o en muy poca cantidad (Schrader et al., 2012). De tal manera, que el obtener ADN de calidad a partir de muestras complejas, es una necesidad creciente para los estudios de genética poblacional de especies silvestres. El presente estudio pretende estandarizar protocolos de extracción y amplificación de ADN, a partir de muestras difíciles obtenidas de segmentos de piel de jaguar (Phantera onca), con el objetivo de colaborar con protocolos reproducibles para la genotipificación de animales a partir de muestras biológicas encontradas u obtenidas de museos o colecciones particulares y contribuir al análisis genético poblacional que pueden ser complementarios a estudios ecológicos, apoyando de esta manera a mejorar las estrategias para su conservación.
Materiales y métodos
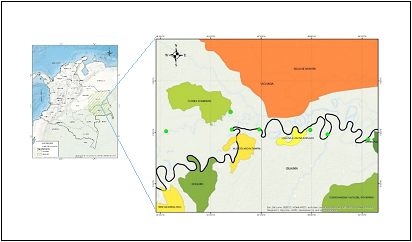
Se utilizaron siete fragmentos de pieles de jaguar, las cuales fueron cedidas por colonos e indígenas de la región de Sapuara- Guaínia, durante un periodo de dos años. Las muestras fueron colectadas y almacenadas a temperatura ambiente por un periodo no menor a 6 años, antes de ser tratadas en el laboratorio de Biología Molecular de la Universidad Nacional Abierta y a Distancia. En la figura No. 1 se muestra la localización de obtención de cada una de las muestras, los puntos verdes corresponden a cada una de las muestras.

Figura No. 1. Localización geográfica de obtención de las pieles
Al momento de la extracción se desconoce el proceso de tratamiento, secado y deshidratación de las muestras, los fragmentos se observan en la figura No. 2. Se recolectaron 6 fragmentos de pieles con indicación del sexo que tuvieron los individuos de los que proceden, que se relacionan en la figura No. 2.

Figura No. 2. Muestras de fragmentos de piel de jaguar (Panthera onca) utilizadas en este estudio
Extracción de ADN
La extracción se realizó en espacios aislados contando con algunas de las medidas de bioseguridad recomendadas (Cooper A, et al. 2000). La extracción y PCR se llevó a cabo en espacios separados utilizando siempre material estéril. Se utilizó como control positivo muestras humanas y como control negativo se realizó todo el proceso, sin utilizar ningún tipo de muestra.
Se probaron tres diferentes protocolos de extracción de ADN: 1- Extracción por fenol cloroformo, 2- extracción con kit comercial Wizard® Genomic DNA Purification Kit Protocol, y 3- extracción de pelos utilizando Chelex-100, con un único proceso de limpieza, hidratación y fragmentación de las pieles. El precipitado final fue corrido en geles de agarosa al 2%. La cantidad y calidad de ADN, se evaluó mediante un espectrofotómetro de marca NanoDrop® ND-1000 Spectrophotometer del Instituto de Genética de la Universidad Nacional de Colombia.
Antes de comenzar el proceso de extracción las pieles fueron limpiadas y cortadas en fragmentos de aproximadamente 2cm x 2 cm y almacenadas en tubos Eppendof, con un bisturí estéril se raspó el interior de la piel, se realizaron tres lavados con agua mili Q estéril y un posterior lavado con etanol al 70%, luego los fragmentos de piel fueron cortados en pequeños segmentos de 0.5 cm x 0.5 cm de longitud, para ser hidratados. El proceso de hidratación consistió en incubar durante 24 horas los fragmentos con 1 ml de buffer TE (Tris- EDTA), luego se centrifugaron y se lavaron con etanol al 70% y se realizó un nuevo lavado con agua mili Q, finalmente se dejaron los fragmentos en buffer TE. Todas las muestras fueron sometidas al mismo proceso de rehidratación.
Con el objetivo determinar la viabilidad del ADN extraido, este fue utilizado para realizar una reacción de PCR, la cual se efectuó utilizando una mezcla de master mix del kit GoTaq® G2 Green Master Mix de Promega, recomendado para muestras forenses y difíciles. Se realizó una reacción de 25 l, se adicionó un volumen de 5 l de ADN y se completó el volumen con de agua ultra pura. Los productos de PCR fueron evaluados en electroforesis en gel de agarosa al 2%.
Se determinaron estadísticos descriptivos como la media y la desviación estándar de los valores: concentración de ADN y pureza de ADN obtenido, para cada uno de los métodos análizados, los resultados fueron realizados utilizando el programa R versión (Copyright (C) 2018 The R Foundation for Statistical Computing), se utilizó la prueba paramétrica de ANOVA para comparar las medias entre los parámetros analizados y se determinó normalidad y homoscedasticidad con las pruebas de Shapiro-Wilk y Barlett.
Resultados y discusión
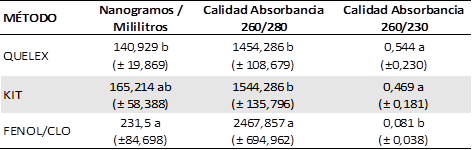
La extracción de ADN de los fragmentos de piel utilizados fue eficiente y se obtuvo ADN de todas las muestran utilizando los tres diferentes tratamientos. Como se esperaba, el método de fenol cloroformo presentó los mejores resultados, en cuanto a la calidad del ADN obtenido (Tabla No. 1). Sin embargo, el mejor tratamiento para la obtención de ADN de calidad y en cantidad, resultó el tratamiento que utilizó un kit comercial de extracción. El tratamiento con resina quelante (Quelex) (Tabla No. 1) utilizado frecuentemente en genética forense, ya que permite obtener ADN a partir de poca cantidad de muestra y con el cual se puede realizar con éxito amplificación de PCR, permitió obtener un fragmento del gen mitocnodrial ATP6-DF3 (Figura No. 3), este método es ampliamente recomendado para casos específicos como el estudio de especies silvestres, en donde se cuenta con poca cantidad de muestra original, como pelos, heces o muestras como las obtenidas en este trabajo de especímenes de museo, ya que permite la amplificación y obtención de segmentos cortos de ADN que pueden ser utilizados en análisis poblacionales.
Tabla No. 1.
Análisis de varianza aplicada en la cantidad y calidad de ADN obtenido en cada uno de los tres tratamientos (Quelex, kit comercial y fenol cloroformo)
Teniendo en cuenta la importancia que determina trabajar con muestras antiguas, se evaluó si existía alguna diferencia significativa entre la edad de la muestra y la calidad de ADN obtenido en cada una de estas. Los resultados indican que no exista diferencia significativa entre la calidad de ADN obtenido y la antigüedad de la misma, medida desde el año en que se referenciaron las muestras. Lo que podría interpretarse como que la calidad de la muestra no sufrió alteración significativa o daños por sufridos por el tiempo en que la muestra fue conservada, luego de la extracción.
Los resultados de la extracción de fenol-cloroformo y de kit se evaluaron en geles de agarosa al 2% p/v, los cuales mostraron una banda de ADN completa, sin evidencias de contaminación por RNA, mostrando poco barrido, lo que podría ser debido a degradación de ADN. La figura No, 3 muestra la amplificación del gen ATP6-DF3, el cual muestra un segmento específico, en control negativo no mostró amplificación.
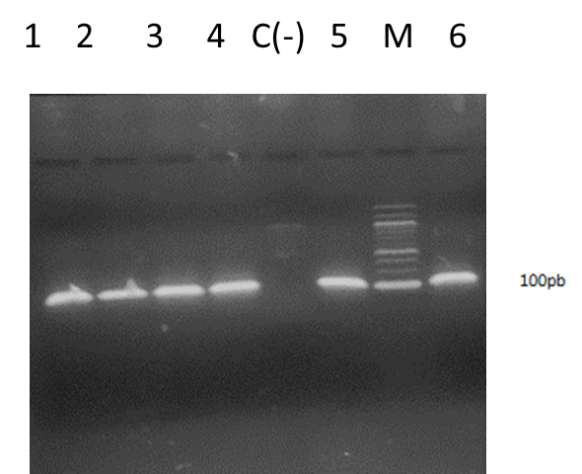
Figura No. 3. Amplificación del gen ATP6-DF3 a partir de ADN extraido con Quelex
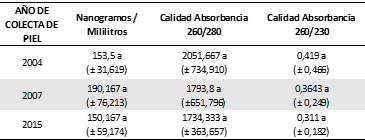
La cuantificación realizada en nanodrop y la determinación de la calidad de ADN, se evaluaron en un equipo nanodrop, mostrando que en todos los tratamientos se obtuvo suficiente cantidad de ADN en y la calidad no afecto la amplicación. Se evaluó si existía alguna correlación entre la edad de la muestra y la cantidad y calidad de ADN obtenido en cada una de estas, los resultados (Tabla No. 2) no permiten concluir que exista correlación entre la cantidad o la calidad de ADN obtenido y la antigüedad de la misma, medida desde el año en que se referenciaron las muestras.
Tabla No. 2
Correlación entre antigüedad de la muestra, cantidad
Conclusiones
El obtener ADN de muestras difíciles dependió de la calidad de los métodos y los reactivos utilizados. En este trabajo se utilizaron protocolos y reactivos utilizados en los laboratorios de genética forense y se lográ obtener ADN en cantidad y calidad suficiente para procedimientos tales como PCR, lo que posibilita la genotipificación de individuos a partir de muestras de pelo y piel. El método de extracción comúnmente utilizado en genética forense (Quelex), el cual es útil para obtener ADN, cuando la cantidad de muestra de la que se extrae el ADN es poca, funciono muy bien en las muestras de pelo obtenidas de los frgamentos de piel de jaguar, estos resultados indican que este puede ser un excelente método para extraer ADN de muestras de cámara trampa u otros dispositivos en donde los animales rasteados dejan material biológico, por lo tanto es altamente recomendable, por ser económico, sencillo y permite la suficiente cantidad de ADN para obtener amplificados de PCR.
Referencias bibliográficas